TRON setzt auf Intel-Server-Cluster im Kampf gegen das Corona-Virus
15.06.2021TRON CoVigator deckt Mutationen im Covid-19 Virus auf
HPC-Cluster analysiert Millionen von Genomproben des SARS-CoV-2-Virus, um Varianten der wichtigsten Krankheitsüberträger zu identifizieren.
Zusammenfassung der Ergebnisse
Die Forscher der TRON gGmbH erforschen seit mehr als zehn Jahren die Immunbiologie von Krebserkrankungen. Als die Covid-19-Pandemie im Jahr 2020 wütete, wendeten die TRON-Wissenschaftler ihre Expertise und ihr Wissen in der Genomanalyse auf das SARS-CoV-2-Virus an. Sie begannen zu untersuchen, wie sich das Virus entwickelt, insbesondere die Varianten des Spike-Glykoproteins, das die Wirtszellen angreift, um den Patienten zu infizieren. Um die Aufgaben der Gensequenzanalyse durchführen zu können, musste TRON seine Rechnerkapazität erweitern. Sie erwarben Intel® Server Systemknoten, die mit Intel Xeon® Scalable Prozessoren der 2. Generation ausgestattet waren, um ihre CoVigator-Genomanalyse-Pipelines auszuführen. Der neue Cluster ermöglichte die Analyse von fast 2 Millionen Virusgenomen und über 30.000 Virusgenom-Sequenzierungsdatensätzen, wobei viele Varianten des Spike-Proteins entdeckt wurden.
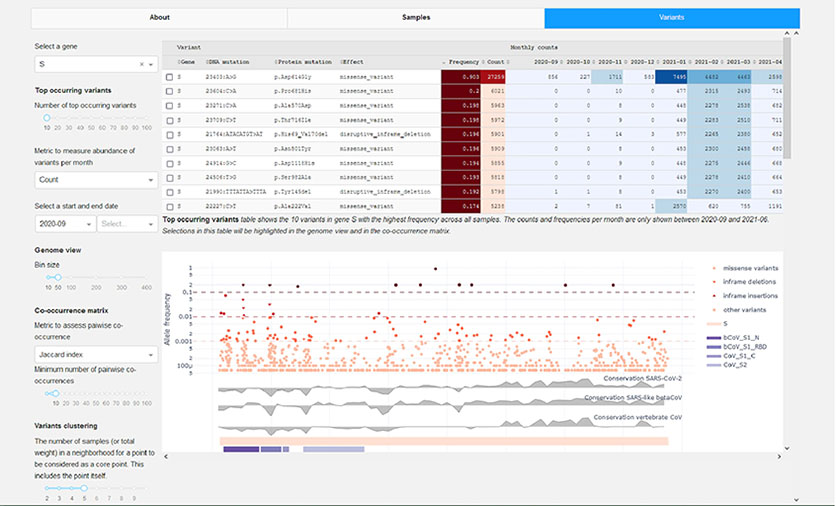
Das CoVigator-Dashboard bietet eine Momentaufnahme der Varianten und anderer wichtiger Daten zum COVID-Virus. (Bild mit freundlicher Genehmigung von Thomas Bukur, TRON)
Herausforderung
TRON ist eine gemeinnützige Forschungseinrichtung, die als unabhängiges Spin-off der Universitätsmedizin der Johannes Gutenberg-Universität Mainz (Deutschland) gegründet wurde. TRON (Abkürzung für Translational Oncology) überbrückt die Lücke zwischen der Grundlagenforschung akademischer Wissenschaftler und den angewandten therapeutischen Lösungen von Pharmaunternehmen. Ihre Forschung ist hauptsächlich auf Krebserkrankungen ausgerichtet.
"In den letzten zehn Jahren lag unser Schwerpunkt auf dem Verständnis der Immunbiologie von Krebserkrankungen", erklärt Martin Löwer, stellvertretender Leiter des Biomarker Development Center (BDC) bei TRON. "Das menschliche Immunsystem kann Tumore erkennen, aber Tumore können sich so entwickeln, dass sie sich dem Immunsystem entziehen. Wir wollten dies untersuchen und verstehen, wie man das Immunsystem so modifizieren und stimulieren kann, dass es Tumore erkennt und neutralisiert, was im Wesentlichen dazu beiträgt, Wege zur Impfung gegen Krebs zu finden."
Die Analyse von Genomen steht im Mittelpunkt des Biomarker Development Center von TRON, wo Tumorgenome und ihre Entwicklung untersucht werden, um nach bestimmten Merkmalen in der DNA, den so genannten Biomarkern, zu suchen. Ein genetischer Biomarker kann das frühe Vorhandensein eines Tumors anzeigen, bevor dieser klinische Symptome zeigt. Molekulare Biomarker können auch eine bestimmte Therapie für einen einzelnen Patienten vorschlagen. Biomarker sind wichtig für das Verständnis der Entwicklung einer Krankheit und dafür, wie eine individualisierte Medizin die Krankheit bei verschiedenen Patienten beeinflussen kann.
„Mit einem neuen Cluster von Servern, die auf Intel® Xeon® Scalable Prozessoren der 2. Generation basieren, analysiert TRON weiterhin bestehende und neue Proben auf virale Mutationen. Die Identifizierung und Aufdeckung der zahlreichen Varianten liefert den Forschern Daten darüber, wie sich das Virus verändert und welche Auswirkungen diese Veränderungen auf Impfstoffe haben könnten.“
Als sich COVID-19 zu einer weltweiten Pandemie entwickelte, wurden Millionen von Menschen rund um den Globus zu Wirten, in denen sich das SARS-CoV-2-Virus durch Mutation weiterentwickelte. Gleichzeitig begannen die Länder mit der Sequenzierung des SARS-CoV-2-Genoms von Tausenden von Patienten und dem Aufbau großer Genomdatensätze für Studien.
Mutationsvarianten einer Krankheit sind für Mediziner und Wissenschaftler von großem Interesse, vor allem, wenn Impfstoffe entwickelt werden, um die Ausbreitung der Krankheit zu bekämpfen. Ähnlich wie bei Krebserkrankungen waren die TRON-Wissenschaftler daran interessiert, wie sich die Entwicklung des SARS-CoV-2-Virus auf die Wirksamkeit der Impfstoffe auswirken könnte, wenn diese auf den Markt kommen. Daher setzten sie ihr Wissen und ihre Erfahrung ein, um das SARS-CoV-2-Genom und insbesondere das Spike-Glykoprotein (Spike-Protein) zu untersuchen, welches das Virus zum Eindringen in menschliche Wirtszellen verwendet.
Die Suche nach Varianten in einem Genom ist ein komplexer Prozess, der viele Berechnungen erfordert. Das SARS-CoV-2-Virus umfasst 30.000 Basenpaare in einer Sequenz (im Vergleich zu 3,2 Milliarden Basenpaaren im menschlichen Genom). Die von Next-Generation-Sequencing (NGS)-Instrumenten gelieferten Sequenzierungsdaten sind eine Zusammenstellung kurzer Abschnitte des gesamten Genoms. Genomdatenspeicher stellen sowohl ganze Genomzusammenstellungen als auch genomische Datensätze bereit, die zunächst ausgerichtet werden müssen, ähnlich wie beim Zusammensetzen von Teilen eines Puzzles mit 30.000 Teilen. Nach der Angleichung werden bei der Analyse die Unterschiede zwischen einem Referenzgenom und der untersuchten Probe untersucht, um die Varianten zu finden und zu markieren. Die Varianten werden weiter gefiltert, um die interessanten Varianten auszuwählen und das so genannte "Rauschen" zu entfernen.
Heutzutage ist das Zusammensetzen, Konvertieren und Analysieren ein hochgradig parallelisierter Prozess, der durch Skalierung auf viele HPC-Server (High Performance Computing) schnell erledigt werden kann. Die Rechenanlagen von TRON werden jedoch ständig von Wissenschaftlern genutzt, um die Krebserkrankungen untersuchen. Um eine Studie über die Spike-Proteinvarianten von SARSCoV-2 abzuschließen und ein Forschungsinstrument für Wissenschaftler auf der ganzen Welt bereitzustellen, wäre mehr Rechenleistung erforderlich, als sie zur Verfügung haben.
Lösung
In Zusammenarbeit mit primeLine Solutions und der Intel Pandemic Response Technology Initiative konnte TRON zehn neue Intel Server System-Knoten erwerben. Diese Knoten sind auf Intel® Xeon® Gold 6240R Prozessoren der 2.Generation mit Intel® SSD S4610 Laufwerken aufgebaut. Der Anmeldeserver verwendet Intel® Xeon® Silver 4208 Prozessoren.
Mit dem neuen System verfügt TRON über 960 dedizierte Threads zur Ausführung der zahlreichen Aufgaben für die Genom-Alignment- und Analyse-Pipelines des Corona Virus Navigator NGS (CoVigator NGS). TRON ist in der Lage, mehr als 20.000 Sequenzierungsdatensätze in weniger als drei Stunden zu analysieren und zu verarbeiten, was eine beinahe Echtzeit-Analyse der ständig wachsenden öffentlich verfügbaren Datensätze ermöglicht.
Die CoVigator-NGS-Pipeline umfasst Trimming, Abgleich, Variantenaufruf und andere Aufgaben. Die TRON-Pipelines umfassen Open-Source-Tools aus vielen genomischen Software-Depots, darunter The Broad Genome Analysis Toolkit (GATK), das für die Intel®-Architektur optimiert wurde, BCFtools, LoFreq und iVar. Die Pipelines sind für andere Wissenschaftler über GitHub verfügbar.
Mit den neuen Rechenkapazitäten begann TRON mit der ersten Untersuchung von 146.917 Genomzusammenstellungen und 2.393 NGS-Datensätzen, die einen Abgleich erforderten, um nichtsynonyme Spike-Protein-Mutationen zu erkennen. Dabei wurden verschiedene Arten von Varianten identifiziert, darunter wiederkehrende gegenüber einzelnen, klonale gegenüber subklonalen und solche, die T-Zell- oder Antikörper-Zielstellen treffen, gegenüber solchen, die diese Ziele nicht treffen. Dies sind alles Varianten, die für Forscher und Therapieentwickler von Nutzen sind.
Die Ergebnisse der Studie zeigen, dass ein bestimmter Prozentsatz von Spike-Protein-Varianten in der gesamten Stichprobenpopulation auftrat (einschließlich vieler Mutationen, die bei einzelnen Patienten gleichzeitig auftraten). Außerdem - und das ist wichtig - zeigt die Studie, dass die Mutationen im Laufe der Zeit zunahmen. Daher ist es von entscheidender Bedeutung, dass die SARS-CoV-2-Genom-Proben weiterhin analysiert und die Auswirkungen der Mutationen auf die Wirksamkeit der Impfstoffe untersucht werden. Die erste Forschungsarbeit von TRON wurde als bioRxiv-Vorabdruck veröffentlicht.
Ergebnis
"Das interessanteste Ergebnis der Forschung", fügt Löwer hinzu, "war, die vielen Varianten des Spike-Proteins zu sehen. Zweitens konnten wir in den letzten anderthalb Jahren genau verfolgen, wie es sich entwickelt hat. Wir sind in der Lage, selbst kleine Veränderungen in der frühen Entwicklungsphase zu erkennen. Wir können sehen, wie es bei einem einzelnen Patienten beginnt und innerhalb des Patienten zu mehreren Varianten und über Populationen und geografische Regionen hinweg mutiert. Und da das Virus weiterhin um die Welt reist, können wir sehen, wie sich die Mutationen im Laufe der Zeit über den Globus bewegen."
Die TRON-Studie war nur der Anfang der Bemühungen, die Evolution des Virus zunächst zu verstehen und dann den Wissenschaftlern zu helfen, sie zu überwachen und zu analysieren. Mit dem neuen Cluster entwickelte TRON eine Plattform zur Erkennung von Mutationen und ein webbasiertes Dashboard, mit dem Wissenschaftler in der Datenbank der Spike-Proteinvarianten navigieren können.
"Als Projekt", so Thomas Bukur von TRON, ein Wissenschaftler, der das CoVigator-Projekt leitet, "wären wir ohne die neuen Server nur in der Lage gewesen, die erste Studie durchzuführen. Aber wir wären nicht in der Lage, eine laufende Studie und einen Service anzubieten, der Millionen von Proben analysiert und Spike-Proteinvarianten identifiziert. Mit dieser Plattform sind wir in der Lage, die Arbeit fortzusetzen."
Der TRON CoVigator-Dienst liefert einen umfassenden Überblick über die zeitliche und räumliche Verteilung der Mutationen des Spike-Proteins des SARS-CoV-2-Virus. Die Pipelines werden wiederholt ausgeführt, um die Datenbank zu aktualisieren. Der CoVigator bietet der Forschungsgemeinschaft ein entscheidendes Instrument, um die Evolution des Virus aufzudecken.
Das Verständnis von Mutationen bei neuen und bekannten Viren ist von entscheidender Bedeutung, um die therapeutische Reaktion auf weit verbreitete und gefährliche Krankheiten anzugehen und kontinuierlich zu steuern. Es wird sicherlich weitere Pandemien geben. Und im Zuge des Klimawandels beobachten Wissenschaftler, dass gefährliche Tropenkrankheiten aus den äquatorialen Regionen in gemäßigtere Zonen wandern. Diese Bedrohungen werden neue Herausforderungen für das Gesundheitswesen mit sich bringen. Die Arbeitsabläufe und Pipelines, die die TRON-Wissenschaftler entwickelt haben, können schnell an neue Viren und Virusstämme angepasst werden und bieten neue Instrumente für die gemeinsame immunbiologische Forschung und Reaktion.
Fazit
TRON wendete sein Fachwissen in der Krebsimmunbiologieforschung auf die Covid-19-Pandemie an, indem es Hunderttausende von SARS-CoV-2-Genom-Proben auf Varianten in seinem Spike-Protein analysierte. Ihre ersten Forschungsergebnisse wurden in einem von bioRxviv vorgedruckten Papier veröffentlicht. Mit einem neuen Cluster von Servern, die auf Intel Xeon Scalable Prozessoren der 2. Generation basieren, analysieren sie weiterhin bestehende und neue Proben auf virale Mutationen. Die Identifizierung und Aufdeckung der zahlreichen Varianten liefert den Forschern Daten darüber, wie sich das Virus verändert und welche Auswirkungen diese Veränderungen auf Impfstoffe haben könnten. TRON stellt sowohl seine Analysepipelines (auf GitHub) als auch die Variantendatenbank über seinen Webdienst TRON CoVigator zur Verfügung.
Verwendete Hardware
- 10 Intel Server System R1208WFTYSR-Rechenknoten
- Intel Xeon 6240R Prozessoren der 2. Generation (480 Kerne und 960 Threads)
- Intel Xeon Silver 4208 Prozessoren der 2. Generation für den Anmeldeknoten
- Intel SSD S4610 480 GB Laufwerke
Englischsprachige PDF als Download